Badacze z Małopolskiego Centrum Biotechnologii przeanalizowali infekcję wirusem SARS-CoV-2 przy użyciu tzw. "nożyc genetycznych", czyli systemu CRISPR-Cas9. Uzyskane wyniki, które zostały opublikowane w "Cells" dostarczają nowych interesujących informacji na temat replikacji wirusa. O szczegółach pisze Aleksandra Synowiec z Virogenetics - laboratorium wirusologii w MCB.
Wirusologia podczas pandemii COVID-19 stała się jedną z najszybciej rozwijających się dziedzin, a wyniki badań mają bezpośrednie przełożenie na sytuację pandemiczną. Poznanie molekularnych mechanizmów infekcji, czyli odpowiedź na pytanie: „Jak wirus SARS-CoV-2 zakaża nasze komórki i jak wywołuje chorobę?”, jest pierwszym krokiem do stworzenia skutecznych leków i strategii terapeutycznych. 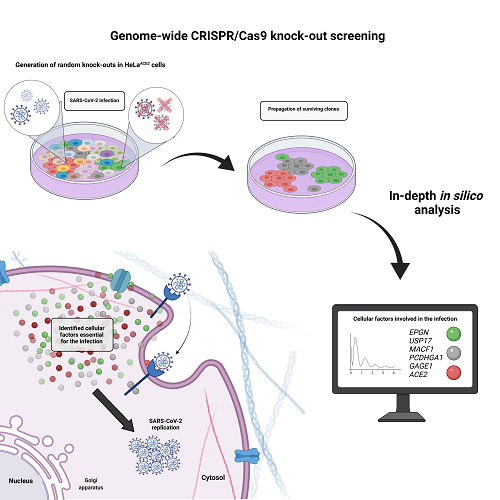
W opublikowanej pracy przy użyciu zaawansowanych metod biologii molekularnej oraz analizy bioinformatycznej, zidentyfikowno nowe czynniki komórkowe, które są potrzebne wirusowi do infekcji. W badaniach została zastosowana metoda CRISPR/Cas9 knock-out screening, która opiera się na technologii CRISPR/Cas9 bazującej na systemie obrony przed mobilnymi elementami genetycznymi u bakterii. Jest to system, w którym białka Cas w połączeniu z krótkimi fragmentami RNA (gRNA; guide RNA) przecinają znajdujące się w komórce sekwencje DNA. Przeniesienie tego systemu obronnego do komórek eukariotycznych (czyli na przykład komórek ludzkich) umożliwia efektywną edycję genów. Technologia ta została wykorzystana do stworzeniu biblioteki knock-out CRISPR/Cas9, w której umieszczono gRNA, które są w stanie „wyłączyć” praktycznie wszystkie znane geny w komórkach ludzkich. Jest to potężne narzędzie umożliwiające identyfikację m.in. interakcji pomiędzy wirusem a komórką.
W ramach opublikowanej pracy przeprowadzono badania w zmodyfikowanej linii komórkowej HeLa, która jest powszechnie używanym modelem do analizy infekcji wirusem SARS-CoV-2. W opisywanych w wynikach udało się zidentyfikować 178 genów, następnie, dokładniejsze analizy pozwoliły wyodrębnić 5 czynników EPGN, USP17, MACF1, PCDHGA1 oraz GAGE1, które wpływają na znaczne ograniczenie (lub opóźnienie w czasie) replikacji wirusa. Ciekawym przykładem jest zidentyfikowane białko MACF1 (ang. microtubule-actin crosslinking factor 1), które bierze udział w wielu procesach oraz ścieżkach sygnałowych związanych z polaryzacją komórki oraz transportem wewnątrzkomórkowym. To białko jest interesujące również z tego względu, że jest zaangażowane w ścieżkę sygnałową Wnt/β-katenin, której rozregulowanie jest obserwowane podczas stanu zapalnego oraz tzw. „sztormu cytokinowego” u pacjentów chorych na COVID-19. Innym interesującym zidentyfikowanym białkiem jest białko EPGN, które jest ligandem dla receptora EGFR pełniącego kluczową rolę m.in. w różnicowaniu komórek. Zostało wykazane, że receptor EGFR jest zaangażowany w replikację SARS-CoV-2.
Aleksandra Synowiec
Małopolskie Centrum Biotechnologii
Artykuł ukazał się w czasopiśmie "Cells" - Synowiec, A.; Jedrysik, M.; Branicki, W.; Klajmon, A.; Lei, J.; Owczarek, K.; Suo, C.; Szczepanski, A.; Wang, J.; Zhang, P.; Labaj, P.P.; Pyrc, K. Identification of Cellular Factors Required for SARS-CoV-2 Replication. Cells 2021, 10, 3159, https://doi.org/10.3390/cells10113159.




